ANALISIS DE LA CALIDAD DEL VINO - Clasificación multiclase
En la primera parte de este análisis enfocamos el problema como aprendizaje supervisado - regresión. El modelo resultante no podemos considerarlo satisfactorio. Vamos a considerar el problema como aprendizaje supervisado - clasificación, concretamente clasificación multiclase.
Carga de datos
Importamos las librerías necesarias:
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.model_selection import train_test_split, GridSearchCV
from sklearn.model_selection import RandomizedSearchCV, cross_val_score
from sklearn.model_selection import cross_validate, cross_val_predict
from sklearn.linear_model import SGDClassifier
from sklearn.dummy import DummyClassifier
from sklearn.naive_bayes import GaussianNB
from sklearn.neighbors import KNeighborsClassifier
from sklearn.ensemble import RandomForestClassifier, AdaBoostClassifier
from sklearn.ensemble import GradientBoostingClassifier
from sklearn.tree import DecisionTreeClassifier, ExtraTreeClassifier
from sklearn import metrics
import xgboost as xgb
%matplotlib inline
import warnings
warnings.filterwarnings('ignore')Leemos los datos y creamos un DataFrame
wine = pd.read_csv("data/wine-quality/winequality-red.csv")Exploración de los datos
wine.head()| fixed acidity | volatile acidity | citric acid | residual sugar | chlorides | free sulfur dioxide | total sulfur dioxide | density | pH | sulphates | alcohol | quality | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 7.4 | 0.70 | 0.00 | 1.9 | 0.076 | 11.0 | 34.0 | 0.9978 | 3.51 | 0.56 | 9.4 | 5 |
| 1 | 7.8 | 0.88 | 0.00 | 2.6 | 0.098 | 25.0 | 67.0 | 0.9968 | 3.20 | 0.68 | 9.8 | 5 |
| 2 | 7.8 | 0.76 | 0.04 | 2.3 | 0.092 | 15.0 | 54.0 | 0.9970 | 3.26 | 0.65 | 9.8 | 5 |
| 3 | 11.2 | 0.28 | 0.56 | 1.9 | 0.075 | 17.0 | 60.0 | 0.9980 | 3.16 | 0.58 | 9.8 | 6 |
| 4 | 7.4 | 0.70 | 0.00 | 1.9 | 0.076 | 11.0 | 34.0 | 0.9978 | 3.51 | 0.56 | 9.4 | 5 |
No vamos a profundizar en la exploración de datos, puesto que ya lo hicimos en la primera parte de este análisis (Calidad del vino - Un problema de regresión).
Preparación de los datos
El único preprocesamiento que vamos a realizar es convertir la variable objetivo "quality" a categórica.
wine["quality_cat"] = wine["quality"].astype("category")wine["quality_cat"].value_counts()5 681
6 638
7 199
4 53
8 18
3 10
Name: quality_cat, dtype: int64
print(f"Porcentaje de cada una de las puntuaciones de calidad")
wine["quality_cat"].value_counts(normalize=True)*100Porcentaje de cada una de las puntuaciones de calidad
5 42.589118
6 39.899937
7 12.445278
4 3.314572
8 1.125704
3 0.625391
Name: quality_cat, dtype: float64
Como ya vimos, el dataset se encuentra significativamente desbalanceado. La mayoría de las instancias (82%) tienen puntuaciones de 6 ó 5.
A continuación creamos el conjunto de predictores y el conjunto con la variable objetivo:
predict_columns = wine.columns[:-2]
predict_columnsIndex(['fixed acidity', 'volatile acidity', 'citric acid', 'residual sugar',
'chlorides', 'free sulfur dioxide', 'total sulfur dioxide', 'density',
'pH', 'sulphates', 'alcohol'],
dtype='object')
X = wine[predict_columns]
y = wine["quality_cat"]Posteriormente, creamos los conjuntos de entrenamiento y prueba, siendo el conjunto de entrenamiento un 80% del dataset completo y el 20% restante el conjunto de prueba:
X_train, X_test, y_train, y_test = train_test_split(X, y,
random_state=42,
test_size=0.2)Línea base
Una pregunta que nos podemos hacer es si está justificado el uso del aprendizaje automático, si nos aporta valor respecto a predecir el azar. Por tanto, lo siguiente que haremos será entrenar un clasificador dummy que utilizaremos como línea base con el que comparar.
En primer lugar, entrenaremos un clasificador que genera predicciones uniformemente al azar.
clf_dummy = DummyClassifier(strategy="uniform", random_state=seed) # Predice al azar
clf_dummy.fit(X_train, y_train)DummyClassifier(random_state=42, strategy='uniform')
cross_val_score(clf_dummy, X_train, y_train, cv=3,
scoring="accuracy", n_jobs=-1).mean()0.16108673901331486
Un clasificador que prediga al azar obtiene una puntuación accuracy del 16%.
Probemos con otro clasificador, pero en este caso, que prediga siempre la clase más frecuente:
clf_dummy = DummyClassifier(strategy="most_frequent", random_state=seed) # Predice siempre la clase más frecuente
clf_dummy.fit(X_train, y_train)DummyClassifier(random_state=42, strategy='most_frequent')
cross_val_score(clf_dummy, X_train, y_train, cv=3, scoring="accuracy", n_jobs=-1).mean()0.4308052321213254
Un clasificador que siempre prediga la clase más frecuente (en nuestro caso la puntuación de calidad 6) obtiene una accuracy del 43%. Vamos a tomar como línea base la predicción de este clasificador dummy.
preds = cross_val_predict(clf_dummy, X_train, y_train, cv=3, n_jobs=-1)Dibujemos su matriz de confusión:
conf_mx = metrics.confusion_matrix(y_train, preds)
conf_mxarray([[ 0, 0, 9, 0, 0, 0],
[ 0, 0, 43, 0, 0, 0],
[ 0, 0, 551, 0, 0, 0],
[ 0, 0, 506, 0, 0, 0],
[ 0, 0, 157, 0, 0, 0],
[ 0, 0, 13, 0, 0, 0]], dtype=int64)
fig = plt.figure(figsize=(8,8))
ax = sns.heatmap(conf_mx, annot=True, fmt="d",
xticklabels=clf_dummy.classes_,
yticklabels=clf_dummy.classes_,)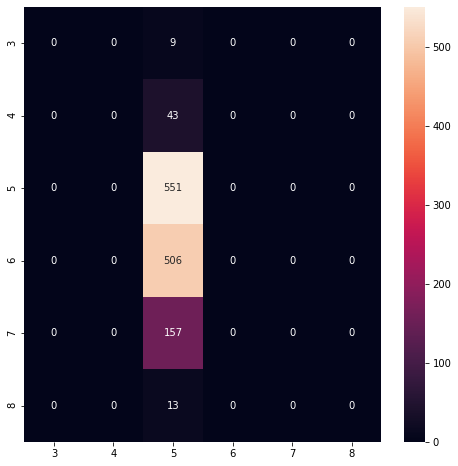
accuracy_base = metrics.accuracy_score(y_train, preds)
precision_base = metrics.precision_score(y_train, preds,
average='weighted',
zero_division=0)
recall_base = metrics.recall_score(y_train, preds,
average='weighted')
f1_base = metrics.f1_score(y_train, preds,
average='weighted')
print(f"Accuracy: {accuracy_base}")
print(f"Precision: {precision_base}")
print(f"Recall: {recall_base}")
print(f"f1: {f1_base}")Accuracy: 0.43080531665363564
Precision: 0.18559322085703928
Recall: 0.43080531665363564
f1: 0.25942484095754453
print(metrics.classification_report(y_train, preds, zero_division=0)) precision recall f1-score support
3 0.00 0.00 0.00 9
4 0.00 0.00 0.00 43
5 0.43 1.00 0.60 551
6 0.00 0.00 0.00 506
7 0.00 0.00 0.00 157
8 0.00 0.00 0.00 13
accuracy 0.43 1279
macro avg 0.07 0.17 0.10 1279
weighted avg 0.19 0.43 0.26 1279
Nuestro clasificador dummy es correcto solo el 19% de las veces (precision) y detecta el 43% de las puntuaciones reales (recall). A menudo es conveniente combinar precisión y sensibilidad en una sola métrica llamada puntuación F1, en particular si necesitamos una forma sencilla de comparar dos clasificadores. La puntuación F1 es la media armónica de precisión y sensibilidad. Mientras que la media regular trata a todos los valores por igual, la media armónica otorga mucho más peso a los valores bajos. Como resultado, el clasificador solo obtendrá una puntuación alta en F1 si tanto la sensibilidad como la precisión son altas. En nuestro caso, F1 = 0,26. Bien, tomemos estas tres métricas como nuestra línea base inicial.
Por tanto, nuestra línea base será:
- Precision: 0.1855
- Recall: 0.4308
- F1: 0.2594
Entrenamiento de diversos modelos
def evaluate_model(estimator, X_train, y_train, cv=5, verbose=True):
"""Print and return cross validation of model
"""
scoring = {"accuracy": "accuracy",
"precision": "precision_weighted",
"recall": "recall_weighted",
"f1": "f1_weighted"}
scores = cross_validate(estimator, X_train, y_train, cv=cv, scoring=scoring)
accuracy, accuracy_std = scores['test_accuracy'].mean(), \
scores['test_accuracy'].std()
precision, precision_std = scores['test_precision'].mean(), \
scores['test_precision'].std()
recall, recall_std = scores['test_recall'].mean(), \
scores['test_recall'].std()
f1, f1_std = scores['test_f1'].mean(), scores['test_f1'].std()
result = {
"Accuracy": accuracy,
"Accuracy std": accuracy_std,
"Precision": precision,
"Precision std": precision_std,
"Recall": recall,
"Recall std": recall_std,
"f1": f1,
"f1 std": f1_std,
}
if verbose:
print(f"Accuracy: {accuracy} - (std: {accuracy_std})")
print(f"Precision: {precision} - (std: {precision_std})")
print(f"Recall: {recall} - (std: {recall_std})")
print(f"f1: {f1} - (std: {f1_std})")
return resultmodels = [GaussianNB(), KNeighborsClassifier(), RandomForestClassifier(random_state=seed),
DecisionTreeClassifier(random_state=seed), ExtraTreeClassifier(random_state=seed),
AdaBoostClassifier(random_state=seed), GradientBoostingClassifier(random_state=seed),
xgb.XGBClassifier()]
model_names = ["Naive Bayes Gaussian", "K Neighbors Classifier", "Random Forest",
"Decision Tree", "Extra Tree", "Ada Boost",
"Gradient Boosting", "XGBoost"]accuracy = []
precision = []
recall = []
f1 = []
for model in range(len(models)):
print(f"Paso {model+1} de {len(models)}")
print(f"...running {model_names[model]}")
clf_scores = evaluate_model(models[model], X_train, y_train)
accuracy.append(clf_scores["Accuracy"])
precision.append(clf_scores["Precision"])
recall.append(clf_scores["Recall"])
f1.append(clf_scores["f1"])Paso 1 de 8
...running Naive Bayes Gaussian
Accuracy: 0.55125 - (std: 0.027102056829233452)
Precision: 0.5646348802130249 - (std: 0.020745595731671666)
Recall: 0.55125 - (std: 0.027102056829233452)
f1: 0.5541082295110215 - (std: 0.023545313928114795)
Paso 2 de 8
...running K Neighbors Classifier
Accuracy: 0.4964828431372549 - (std: 0.013777320430796238)
Precision: 0.472985448646598 - (std: 0.015072330289309464)
Recall: 0.4964828431372549 - (std: 0.013777320430796238)
f1: 0.4749703234382818 - (std: 0.01350721905804416)
Paso 3 de 8
...running Random Forest
Accuracy: 0.6826194852941176 - (std: 0.03746156433885403)
Precision: 0.6585977991402794 - (std: 0.0406774341137893)
Recall: 0.6826194852941176 - (std: 0.03746156433885403)
f1: 0.6642629277794576 - (std: 0.03850557708999431)
Paso 4 de 8
...running Decision Tree
Accuracy: 0.6012714460784314 - (std: 0.028539445741031087)
Precision: 0.5978218408820158 - (std: 0.025874687130953537)
Recall: 0.6012714460784314 - (std: 0.028539445741031087)
f1: 0.5978989958450711 - (std: 0.0264307770802976)
Paso 5 de 8
...running Extra Tree
Accuracy: 0.5676348039215686 - (std: 0.032774267548303905)
Precision: 0.5697402861119303 - (std: 0.030789932683965727)
Recall: 0.5676348039215686 - (std: 0.032774267548303905)
f1: 0.5668315018481278 - (std: 0.031722387303563124)
Paso 6 de 8
...running Ada Boost
Accuracy: 0.5504748774509804 - (std: 0.03954230035312734)
Precision: 0.48457698009594374 - (std: 0.05118366184736229)
Recall: 0.5504748774509804 - (std: 0.03954230035312734)
f1: 0.5052214324230416 - (std: 0.03764434709325329)
Paso 7 de 8
...running Gradient Boosting
Accuracy: 0.6474325980392157 - (std: 0.03472028817662461)
Precision: 0.6218203966653049 - (std: 0.03370831758409691)
Recall: 0.6474325980392157 - (std: 0.03472028817662461)
f1: 0.6328837599218248 - (std: 0.03442412231869498)
Paso 8 de 8
...running XGBoost
[15:57:22] WARNING: C:/Users/Administrator/workspace/xgboost-win64_release_1.4.0/src/learner.cc:1095: Starting in XGBoost 1.3.0, the default evaluation metric used with the objective 'multi:softprob' was changed from 'merror' to 'mlogloss'. Explicitly set eval_metric if you'd like to restore the old behavior.
[15:57:22] WARNING: C:/Users/Administrator/workspace/xgboost-win64_release_1.4.0/src/learner.cc:1095: Starting in XGBoost 1.3.0, the default evaluation metric used with the objective 'multi:softprob' was changed from 'merror' to 'mlogloss'. Explicitly set eval_metric if you'd like to restore the old behavior.
[15:57:23] WARNING: C:/Users/Administrator/workspace/xgboost-win64_release_1.4.0/src/learner.cc:1095: Starting in XGBoost 1.3.0, the default evaluation metric used with the objective 'multi:softprob' was changed from 'merror' to 'mlogloss'. Explicitly set eval_metric if you'd like to restore the old behavior.
[15:57:23] WARNING: C:/Users/Administrator/workspace/xgboost-win64_release_1.4.0/src/learner.cc:1095: Starting in XGBoost 1.3.0, the default evaluation metric used with the objective 'multi:softprob' was changed from 'merror' to 'mlogloss'. Explicitly set eval_metric if you'd like to restore the old behavior.
[15:57:24] WARNING: C:/Users/Administrator/workspace/xgboost-win64_release_1.4.0/src/learner.cc:1095: Starting in XGBoost 1.3.0, the default evaluation metric used with the objective 'multi:softprob' was changed from 'merror' to 'mlogloss'. Explicitly set eval_metric if you'd like to restore the old behavior.
Accuracy: 0.6560079656862745 - (std: 0.023339659857252816)
Precision: 0.6346626310195044 - (std: 0.028312439862179448)
Recall: 0.6560079656862745 - (std: 0.023339659857252816)
f1: 0.6420686275076488 - (std: 0.024663282704859676)
df_result = pd.DataFrame({"Model": model_names,
"accuracy": accuracy,
"precision": precision,
"recall": recall,
"f1": f1})
df_result.sort_values(by="f1", ascending=False)| Model | accuracy | precision | recall | f1 | |
|---|---|---|---|---|---|
| 2 | Random Forest | 0.682619 | 0.658598 | 0.682619 | 0.664263 |
| 7 | XGBoost | 0.656008 | 0.634663 | 0.656008 | 0.642069 |
| 6 | Gradient Boosting | 0.647433 | 0.621820 | 0.647433 | 0.632884 |
| 3 | Decision Tree | 0.601271 | 0.597822 | 0.601271 | 0.597899 |
| 4 | Extra Tree | 0.567635 | 0.569740 | 0.567635 | 0.566832 |
| 0 | Naive Bayes Gaussian | 0.551250 | 0.564635 | 0.551250 | 0.554108 |
| 5 | Ada Boost | 0.550475 | 0.484577 | 0.550475 | 0.505221 |
| 1 | K Neighbors Classifier | 0.496483 | 0.472985 | 0.496483 | 0.474970 |
Vamos a visualizar la comparativa de los diferentes modelos / métricas:
metrics_list = ["f1", "accuracy", "precision", "recall"]
for metric in metrics_list:
df_result.sort_values(by=metric).plot.barh("Model", metric)
plt.title(f"Model by {metric}")
plt.show()



Obtenemos que el modelo que tiene mejor rendimiento es Random Forest. Examinemos un poco más en detalle la ejecución de Random Forest:
clf_rf = RandomForestClassifier(random_state=seed)
preds = cross_val_predict(clf_rf, X_train, y_train, cv=5, n_jobs=-1)clf_rf.get_params(){'bootstrap': True,
'ccp_alpha': 0.0,
'class_weight': None,
'criterion': 'gini',
'max_depth': None,
'max_features': 'auto',
'max_leaf_nodes': None,
'max_samples': None,
'min_impurity_decrease': 0.0,
'min_impurity_split': None,
'min_samples_leaf': 1,
'min_samples_split': 2,
'min_weight_fraction_leaf': 0.0,
'n_estimators': 100,
'n_jobs': None,
'oob_score': False,
'random_state': 42,
'verbose': 0,
'warm_start': False}
pd.crosstab(y_train, preds, rownames = ['Actual'], colnames =['Predicción'])| Predicción | 3 | 4 | 5 | 6 | 7 | 8 |
|---|---|---|---|---|---|---|
| Actual | ||||||
| 3 | 0 | 1 | 7 | 1 | 0 | 0 |
| 4 | 1 | 0 | 32 | 9 | 1 | 0 |
| 5 | 0 | 2 | 434 | 108 | 7 | 0 |
| 6 | 0 | 1 | 116 | 364 | 25 | 0 |
| 7 | 0 | 0 | 14 | 70 | 73 | 0 |
| 8 | 0 | 0 | 0 | 6 | 5 | 2 |
print(metrics.classification_report(y_train, preds, zero_division=0)) precision recall f1-score support
3 0.00 0.00 0.00 9
4 0.00 0.00 0.00 43
5 0.72 0.79 0.75 551
6 0.65 0.72 0.68 506
7 0.66 0.46 0.54 157
8 1.00 0.15 0.27 13
accuracy 0.68 1279
macro avg 0.50 0.35 0.37 1279
weighted avg 0.66 0.68 0.66 1279
El modelo es correcto el 66% de las veces (precision) y detecta el 68% de las puntuaciones reales (recall). Siendo la puntuación F1 de 0,66. Bueno, ha mejorado significativamente nuestra línea base (recordemos, precision=19%, recall=43% y F1=0,26).
El % de mejora del indicador F1 respecto a la línea base es:
% diferencia F1= (0.66 - 0.26) / 0.66 * 100 = 60.6%
Realmente la mejora respecto a la línea base es considerable, un 60%. Podemos concluir que está justificado el uso de aprendizaje automático para predecir la puntuación de calidad del vino.
En general, si el porcentaje de mejora respecto a nuestra línea base no es mayor que un 5% deberíamos reconsiderar el uso de aprendizaje automático.
Al examinar en detalle el resultado de las predicciones, podemos observar que es pésimo en las puntuaciones extremas (3, 4 y 8) y bastante malo en la puntuación 7.
Ajuste fino de hiperparámetros
Vamos a realizar un ajuste de hiperparámetros a ver si se consigue alguna mejora.
param_grid = [
{"n_estimators": range(20, 200, 20),
"bootstrap": [True, False],
"criterion": ["gini", "entropy"],
"max_depth": [2, 4, 6, 8, 10, 12, 14, None],
"max_features": ["auto", "sqrt", "log2"],
"min_samples_split": [2, 5, 10],
"min_samples_leaf": [1, 2, 4],
}
]
clf_rf = RandomForestClassifier(random_state=seed, n_jobs=-1)Ajuste inicial con Randomize Search
En primer lugar hacemos un barrido rápido aleatorio:
clf_random = RandomizedSearchCV(clf_rf, param_grid, n_iter = 200, cv = 5,
scoring="f1_weighted", verbose=2,
random_state=seed, n_jobs = -1)clf_random.fit(X_train, y_train)Fitting 5 folds for each of 200 candidates, totalling 1000 fits
RandomizedSearchCV(cv=5,
estimator=RandomForestClassifier(n_jobs=-1, random_state=42),
n_iter=200, n_jobs=-1,
param_distributions=[{'bootstrap': [True, False],
'criterion': ['gini', 'entropy'],
'max_depth': [2, 4, 6, 8, 10, 12, 14,
None],
'max_features': ['auto', 'sqrt',
'log2'],
'min_samples_leaf': [1, 2, 4],
'min_samples_split': [2, 5, 10],
'n_estimators': range(20, 200, 20)}],
random_state=42, scoring='f1_weighted', verbose=2)
clf_random.best_params_{'n_estimators': 40,
'min_samples_split': 2,
'min_samples_leaf': 2,
'max_features': 'sqrt',
'max_depth': 10,
'criterion': 'entropy',
'bootstrap': False}
preds = cross_val_predict(clf_random.best_estimator_,
X_train, y_train,
cv=5, n_jobs=-1)
print(metrics.classification_report(y_train, preds, zero_division=0)) precision recall f1-score support
3 0.00 0.00 0.00 9
4 0.00 0.00 0.00 43
5 0.72 0.81 0.76 551
6 0.66 0.71 0.68 506
7 0.66 0.46 0.55 157
8 1.00 0.15 0.27 13
accuracy 0.69 1279
macro avg 0.51 0.36 0.38 1279
weighted avg 0.66 0.69 0.67 1279
Ajuste final con GridSearch
Proseguimos con un ajuste final usando GridSearch:
param_grid = [
{"n_estimators": range(130, 200, 10),
"bootstrap": [True, False],
"criterion": ["gini", "entropy"],
"max_depth": [2, 4, 6, 8, 10, 12, 14, None],
"max_features": ["auto", "sqrt", "log2"],
"min_samples_split": [2, 5, 10],
"min_samples_leaf": [1, 2, 4],
}
]
clf_rf = RandomForestClassifier(random_state=seed, n_jobs=-1)grid_search = GridSearchCV(clf_rf, param_grid, cv=5,
scoring="f1_weighted", verbose=2, n_jobs=-1)grid_search.fit(X_train, y_train)Fitting 5 folds for each of 6048 candidates, totalling 30240 fits
GridSearchCV(cv=5, estimator=RandomForestClassifier(n_jobs=-1, random_state=42),
n_jobs=-1,
param_grid=[{'bootstrap': [True, False],
'criterion': ['gini', 'entropy'],
'max_depth': [2, 4, 6, 8, 10, 12, 14, None],
'max_features': ['auto', 'sqrt', 'log2'],
'min_samples_leaf': [1, 2, 4],
'min_samples_split': [2, 5, 10],
'n_estimators': range(130, 200, 10)}],
scoring='f1_weighted', verbose=2)
grid_search.best_params_{'bootstrap': True,
'criterion': 'entropy',
'max_depth': 10,
'max_features': 'auto',
'min_samples_leaf': 1,
'min_samples_split': 2,
'n_estimators': 170}
final_model = grid_search.best_estimator_
preds = cross_val_predict(final_model, X_train, y_train, cv=5, n_jobs=-1)pd.crosstab(y_train, preds, rownames = ['Actual'], colnames =['Predicción'])| Predicción | 3 | 4 | 5 | 6 | 7 | 8 |
|---|---|---|---|---|---|---|
| Actual | ||||||
| 3 | 0 | 1 | 6 | 2 | 0 | 0 |
| 4 | 1 | 0 | 31 | 10 | 1 | 0 |
| 5 | 0 | 0 | 451 | 94 | 6 | 0 |
| 6 | 0 | 0 | 113 | 365 | 28 | 0 |
| 7 | 0 | 0 | 10 | 78 | 69 | 0 |
| 8 | 0 | 0 | 0 | 6 | 5 | 2 |
print(metrics.classification_report(y_train, preds)) precision recall f1-score support
3 0.00 0.00 0.00 9
4 0.00 0.00 0.00 43
5 0.74 0.82 0.78 551
6 0.66 0.72 0.69 506
7 0.63 0.44 0.52 157
8 1.00 0.15 0.27 13
accuracy 0.69 1279
macro avg 0.50 0.36 0.37 1279
weighted avg 0.67 0.69 0.67 1279
Tras el ajuste de hiperparámetros se consigue una muy ligera mejora respecto a los hiperparámetros por defecto. Es correcto el 67% de las veces (precision) y detecta el 69% de las puntuaciones reales (recall). Siendo la puntuación F1 de 0,67. Lo que mejora significativamente nuestra línea base (recordemos, precision=19%, recall=43% y F1=0,26).
Por último veamos cómo se ejecuta en el conjunto de prueba:
y_pred = final_model.predict(X_test)pd.crosstab(y_test, y_pred, rownames = ['Actual'], colnames =['Predicción'])| Predicción | 5 | 6 | 7 | 8 |
|---|---|---|---|---|
| Actual | ||||
| 3 | 1 | 0 | 0 | 0 |
| 4 | 6 | 4 | 0 | 0 |
| 5 | 101 | 28 | 1 | 0 |
| 6 | 37 | 89 | 6 | 0 |
| 7 | 0 | 22 | 19 | 1 |
| 8 | 0 | 1 | 4 | 0 |
print(metrics.classification_report(y_test, y_pred, zero_division=0)) precision recall f1-score support
3 0.00 0.00 0.00 1
4 0.00 0.00 0.00 10
5 0.70 0.78 0.73 130
6 0.62 0.67 0.64 132
7 0.63 0.45 0.53 42
8 0.00 0.00 0.00 5
accuracy 0.65 320
macro avg 0.32 0.32 0.32 320
weighted avg 0.62 0.65 0.63 320
Es correcto el 62% de las veces (precision) y detecta el 65% de las puntuaciones reales (recall). Siendo la puntuación F1 de 0,65.
accuracy_best = metrics.accuracy_score(y_test, y_pred)
precision_best = metrics.precision_score(y_test, y_pred,
average='weighted',
zero_division=0)
recall_best = metrics.recall_score(y_test, y_pred,
average='weighted')
f1_best = metrics.f1_score(y_test, y_pred,
average='weighted')Matriz de confusión
conf_mx = metrics.confusion_matrix(y_test, y_pred)fig = plt.figure(figsize=(8,8))
ax = sns.heatmap(conf_mx, annot=True, fmt="d",
xticklabels=final_model.classes_,
yticklabels=final_model.classes_,)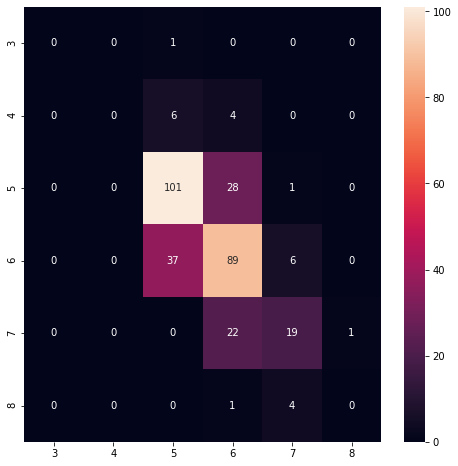
Feature importances
feature_importances = final_model.feature_importances_
feature_importancesarray([0.06970454, 0.10304422, 0.07397403, 0.06774786, 0.07530372,
0.06051697, 0.09785917, 0.0830556 , 0.06881937, 0.12760515,
0.17236938])
sorted(zip(feature_importances, X_test.columns), reverse=True)[(0.17236937962448678, 'alcohol'),
(0.12760514906291182, 'sulphates'),
(0.10304421805642286, 'volatile acidity'),
(0.09785917335424621, 'total sulfur dioxide'),
(0.0830555965951595, 'density'),
(0.0753037227200391, 'chlorides'),
(0.07397402652373279, 'citric acid'),
(0.06970454021889655, 'fixed acidity'),
(0.06881936733049614, 'pH'),
(0.06774786106526597, 'residual sugar'),
(0.06051696544834242, 'free sulfur dioxide')]
feature_imp = pd.Series(feature_importances, index=X_train.columns).sort_values(ascending=False)
feature_imp.plot(kind='bar')
plt.title('Feature Importances');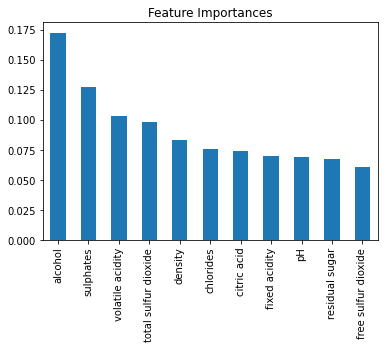
Observamos que las características que más influencia tienen en nuestro modelo son alcohol y sulphates, seguidas por volatile acidity y total sulfur dioxide.
Selección de características
Vamos a usar RFECV para determinar el nº de características válidas con cross-validation.
from sklearn.feature_selection import RFECV
from sklearn.model_selection import StratifiedKFold
selector = RFECV(final_model, step=1, cv=StratifiedKFold())
selector = selector.fit(X_train, y_train)
pd.DataFrame({"Feature": predict_columns, "Support": selector.support_})| Feature | Support | |
|---|---|---|
| 0 | fixed acidity | True |
| 1 | volatile acidity | True |
| 2 | citric acid | True |
| 3 | residual sugar | True |
| 4 | chlorides | True |
| 5 | free sulfur dioxide | True |
| 6 | total sulfur dioxide | True |
| 7 | density | True |
| 8 | pH | True |
| 9 | sulphates | True |
| 10 | alcohol | True |
pd.DataFrame({"Feature": predict_columns, "Ranking": selector.ranking_})| Feature | Ranking | |
|---|---|---|
| 0 | fixed acidity | 1 |
| 1 | volatile acidity | 1 |
| 2 | citric acid | 1 |
| 3 | residual sugar | 1 |
| 4 | chlorides | 1 |
| 5 | free sulfur dioxide | 1 |
| 6 | total sulfur dioxide | 1 |
| 7 | density | 1 |
| 8 | pH | 1 |
| 9 | sulphates | 1 |
| 10 | alcohol | 1 |
# Dibuja el número de features vs la puntuación a través de cross-validation
plt.figure()
plt.xlabel("Nº de features seleccionadas")
plt.ylabel("Puntuación cross validation")
plt.plot(range(1, len(selector.grid_scores_) + 1), selector.grid_scores_)
plt.show()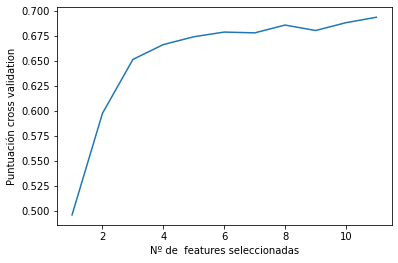
Observando la gráfica se concluye que todas las variables son importantes para el modelo, ya que se obtiene la máxima puntuación con las 10 características seleccionadas.
selector.grid_scores_array([0.49564951, 0.59737132, 0.65132353, 0.6661826 , 0.6739951 ,
0.67869792, 0.67790135, 0.68573223, 0.68025123, 0.68808211,
0.69354167])
Guardado del modelo
Por último, guardamos nuestro modelo entrenado para futuras predicciones.
import joblib
joblib.dump(final_model, "final_model_clf.joblib", compress=True)
#final_model = joblib.load("final_model_clf.joblib")['final_model_clf.joblib']
Comentarios finales a los resultados
Nuestra línea de base de partida, obtenida a partir de un clasificador que siempre predice la clase más frecuente, es la siguiente:
- Precision: 19%
- Recall: 43%
- Accuracy: 43%
- f1: 0.26
Una vez entrenados diversos modelos, el que mejores resultados ha proporcionados es RandomForest. Después de realizar un ajuste fino de hiperparámetros obtenemos las siguientes métricas:
- Precision: 67%
- Recall: 69%
- Accuracy: 69%
- f1: 0.67
La evaluación en el conjunto de prueba es la siguiente:
- Precision: 62%
- Recall: 65%
- Accuracy: 65%
- f1: 0.63
Al ser multiclase, estamos hablando de puntuaciones ponderadas. Sin embargo, las puntuaciones obtenidas por cada clase son muy dispares. Se puede observar que el resultado es pésimo en las puntuaciones extremas (3, 4 y 8). Según vimos en la distribución de la variable objetivo, ésta se encuentra muy desbalanceada, apenas existen observaciones para los valores extremos, por lo que el modelo no tiene suficientes datos de entrenamiento para todas las puntuaciones de calidad.
Todas las variables predictoras son relevantes para el modelo. Las tres que más afectan en la predicción son las siguientes:
- alcohol
- sulphates
- volatile acidity.
Podría ser interesante evaluar el modelo segmentando nuestra variable objetivo en rangos de calidad (por ejemplo, baja, media y alta) y comprobar si obtenemos mejores resultados.