En este post repasaremos las principales fases que componen un proyecto de Machine Learning.
Existen ocho pasos principales:
-
Encuadrar el problema y disponer de la visión global.
-
Obtener los datos.
-
Explorar los datos para obtener ideas.
-
Preparar los datos para exponer lo mejor posible los patrones de datos subyacentes a los algoritmos de Machine Learning.
-
Explorar muchos modelos diferentes y preseleccionar los mejores.
-
Afinar nuestros modelos y combinarlos en una gran solución.
-
Presentar nuestra solución.
-
Implantar, monitorizar y mantener nuestro sistema.
Disponemos un conjunto de datos que contiene diversas características de variantes de tinto y blanco del vino portugués “Vinho Verde”. Disponemos de variables químicas, como son la cantidad de alcohol, ácido cítrico, acidez, densidad, pH, etc; así como de una variable sensorial y subjetiva como es la puntuación con la que un grupo de expertos calificaron la calidad del vino: entre 0 (muy malo) y 10 (muy excelente).
El objetivo es desarrollar un modelo que pueda predecir la puntuación de calidad dados dichos indicadores bioquímicos.
Lo primero que nos viene a la mente son una serie de preguntas básicas:
-
¿Cómo se enmarcaría este problema (supervisado, no supervisado, etc.)?
-
¿Cuál es la variable objetivo? ¿Cuáles son los predictores?
-
¿Cómo vamos a medir el rendimiento de nuestro modelo?

El codigo python utilizado en este artículo está disponible en mi repositorio github
En primer lugar importamos todas las librerías necesarias:
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.model_selection import train_test_split, GridSearchCV, cross_val_score, cross_validate
from sklearn.linear_model import LinearRegression, SGDRegressor, Lasso, ElasticNet, Ridge
from sklearn.dummy import DummyRegressor
from sklearn.ensemble import RandomForestRegressor, ExtraTreesRegressor
from sklearn.svm import SVR
from sklearn import metrics
%matplotlib inlineObtención de los datos
El dataset se encuentra igualmente disponible en Kaggle o en UCI
Podemos cargarlo directamente desde la url o una vez descargado desde nuestra carpeta data.
red = pd.read_csv("data/wine-quality/winequality-red.csv")Verificamos el tamaño y el tipo de los datos
red.shape(1599, 12)
red.head()| fixed acidity | volatile acidity | citric acid | residual sugar | chlorides | free sulfur dioxide | total sulfur dioxide | density | pH | sulphates | alcohol | quality | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 7.4 | 0.70 | 0.00 | 1.9 | 0.076 | 11.0 | 34.0 | 0.9978 | 3.51 | 0.56 | 9.4 | 5 |
| 1 | 7.8 | 0.88 | 0.00 | 2.6 | 0.098 | 25.0 | 67.0 | 0.9968 | 3.20 | 0.68 | 9.8 | 5 |
| 2 | 7.8 | 0.76 | 0.04 | 2.3 | 0.092 | 15.0 | 54.0 | 0.9970 | 3.26 | 0.65 | 9.8 | 5 |
| 3 | 11.2 | 0.28 | 0.56 | 1.9 | 0.075 | 17.0 | 60.0 | 0.9980 | 3.16 | 0.58 | 9.8 | 6 |
| 4 | 7.4 | 0.70 | 0.00 | 1.9 | 0.076 | 11.0 | 34.0 | 0.9978 | 3.51 | 0.56 | 9.4 | 5 |
red.info()<class 'pandas.core.frame.DataFrame'>
RangeIndex: 1599 entries, 0 to 1598
Data columns (total 12 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 fixed acidity 1599 non-null float64
1 volatile acidity 1599 non-null float64
2 citric acid 1599 non-null float64
3 residual sugar 1599 non-null float64
4 chlorides 1599 non-null float64
5 free sulfur dioxide 1599 non-null float64
6 total sulfur dioxide 1599 non-null float64
7 density 1599 non-null float64
8 pH 1599 non-null float64
9 sulphates 1599 non-null float64
10 alcohol 1599 non-null float64
11 quality 1599 non-null int64
dtypes: float64(11), int64(1)
memory usage: 150.0 KB
Realizamos una serie de comprobaciones para conocer la naturaleza de los datos con los que vamos a trabajar: tipo, valores únicos, número de valores únicos y su porcentaje, valores medios y desviación estándar.
pd.DataFrame({"Type": red.dtypes,
"Unique": red.nunique(),
"Null": red.isnull().sum(),
"Null percent": red.isnull().sum() / len(red),
"Mean": red.mean(),
"Std": red.std()})| Type | Unique | Null | Null percent | Mean | Std | |
|---|---|---|---|---|---|---|
| fixed acidity | float64 | 96 | 0 | 0.0 | 8.319637 | 1.741096 |
| volatile acidity | float64 | 143 | 0 | 0.0 | 0.527821 | 0.179060 |
| citric acid | float64 | 80 | 0 | 0.0 | 0.270976 | 0.194801 |
| residual sugar | float64 | 91 | 0 | 0.0 | 2.538806 | 1.409928 |
| chlorides | float64 | 153 | 0 | 0.0 | 0.087467 | 0.047065 |
| free sulfur dioxide | float64 | 60 | 0 | 0.0 | 15.874922 | 10.460157 |
| total sulfur dioxide | float64 | 144 | 0 | 0.0 | 46.467792 | 32.895324 |
| density | float64 | 436 | 0 | 0.0 | 0.996747 | 0.001887 |
| pH | float64 | 89 | 0 | 0.0 | 3.311113 | 0.154386 |
| sulphates | float64 | 96 | 0 | 0.0 | 0.658149 | 0.169507 |
| alcohol | float64 | 65 | 0 | 0.0 | 10.422983 | 1.065668 |
| quality | int64 | 6 | 0 | 0.0 | 5.636023 | 0.807569 |
Mmmmm, no existen valores nulos, ¡qué buen dataset!
red.describe().T| count | mean | std | min | 25% | 50% | 75% | max | |
|---|---|---|---|---|---|---|---|---|
| fixed acidity | 1599.0 | 8.319637 | 1.741096 | 4.60000 | 7.1000 | 7.90000 | 9.200000 | 15.90000 |
| volatile acidity | 1599.0 | 0.527821 | 0.179060 | 0.12000 | 0.3900 | 0.52000 | 0.640000 | 1.58000 |
| citric acid | 1599.0 | 0.270976 | 0.194801 | 0.00000 | 0.0900 | 0.26000 | 0.420000 | 1.00000 |
| residual sugar | 1599.0 | 2.538806 | 1.409928 | 0.90000 | 1.9000 | 2.20000 | 2.600000 | 15.50000 |
| chlorides | 1599.0 | 0.087467 | 0.047065 | 0.01200 | 0.0700 | 0.07900 | 0.090000 | 0.61100 |
| free sulfur dioxide | 1599.0 | 15.874922 | 10.460157 | 1.00000 | 7.0000 | 14.00000 | 21.000000 | 72.00000 |
| total sulfur dioxide | 1599.0 | 46.467792 | 32.895324 | 6.00000 | 22.0000 | 38.00000 | 62.000000 | 289.00000 |
| density | 1599.0 | 0.996747 | 0.001887 | 0.99007 | 0.9956 | 0.99675 | 0.997835 | 1.00369 |
| pH | 1599.0 | 3.311113 | 0.154386 | 2.74000 | 3.2100 | 3.31000 | 3.400000 | 4.01000 |
| sulphates | 1599.0 | 0.658149 | 0.169507 | 0.33000 | 0.5500 | 0.62000 | 0.730000 | 2.00000 |
| alcohol | 1599.0 | 10.422983 | 1.065668 | 8.40000 | 9.5000 | 10.20000 | 11.100000 | 14.90000 |
| quality | 1599.0 | 5.636023 | 0.807569 | 3.00000 | 5.0000 | 6.00000 | 6.000000 | 8.00000 |
Exploración de los datos
El siguiente paso será realizar un análisis exploratorio de los datos. ¿Cómo se distribuyen las características?
red.hist(bins=50, figsize=(15,12));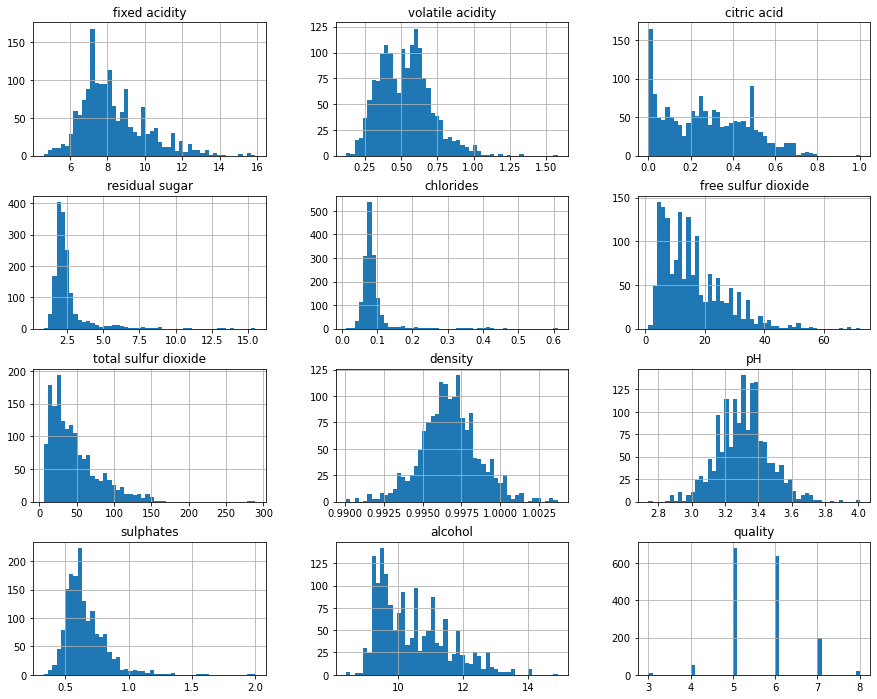
Verifiquemos ahora cómo se distribuye nuestra variable objetivo (la puntuación de calidad):
print(f"Percentage of quality scores")
red["quality"].value_counts(normalize=True) * 100Percentage of quality scores
5 42.589118
6 39.899937
7 12.445278
4 3.314572
8 1.125704
3 0.625391
Name: quality, dtype: float64
Podemos comprobar que se encuentra significativamente desbalanceada. La mayoría de las instancias (82%) tienen puntuaciones de 5 ó 6.
Vamos a verificar las correlaciones entre las características del dataset:
corr_matrix = red.corr()
corr_matrix| fixed acidity | volatile acidity | citric acid | residual sugar | chlorides | free sulfur dioxide | total sulfur dioxide | density | pH | sulphates | alcohol | quality | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| fixed acidity | 1.000000 | -0.256131 | 0.671703 | 0.114777 | 0.093705 | -0.153794 | -0.113181 | 0.668047 | -0.682978 | 0.183006 | -0.061668 | 0.124052 |
| volatile acidity | -0.256131 | 1.000000 | -0.552496 | 0.001918 | 0.061298 | -0.010504 | 0.076470 | 0.022026 | 0.234937 | -0.260987 | -0.202288 | -0.390558 |
| citric acid | 0.671703 | -0.552496 | 1.000000 | 0.143577 | 0.203823 | -0.060978 | 0.035533 | 0.364947 | -0.541904 | 0.312770 | 0.109903 | 0.226373 |
| residual sugar | 0.114777 | 0.001918 | 0.143577 | 1.000000 | 0.055610 | 0.187049 | 0.203028 | 0.355283 | -0.085652 | 0.005527 | 0.042075 | 0.013732 |
| chlorides | 0.093705 | 0.061298 | 0.203823 | 0.055610 | 1.000000 | 0.005562 | 0.047400 | 0.200632 | -0.265026 | 0.371260 | -0.221141 | -0.128907 |
| free sulfur dioxide | -0.153794 | -0.010504 | -0.060978 | 0.187049 | 0.005562 | 1.000000 | 0.667666 | -0.021946 | 0.070377 | 0.051658 | -0.069408 | -0.050656 |
| total sulfur dioxide | -0.113181 | 0.076470 | 0.035533 | 0.203028 | 0.047400 | 0.667666 | 1.000000 | 0.071269 | -0.066495 | 0.042947 | -0.205654 | -0.185100 |
| density | 0.668047 | 0.022026 | 0.364947 | 0.355283 | 0.200632 | -0.021946 | 0.071269 | 1.000000 | -0.341699 | 0.148506 | -0.496180 | -0.174919 |
| pH | -0.682978 | 0.234937 | -0.541904 | -0.085652 | -0.265026 | 0.070377 | -0.066495 | -0.341699 | 1.000000 | -0.196648 | 0.205633 | -0.057731 |
| sulphates | 0.183006 | -0.260987 | 0.312770 | 0.005527 | 0.371260 | 0.051658 | 0.042947 | 0.148506 | -0.196648 | 1.000000 | 0.093595 | 0.251397 |
| alcohol | -0.061668 | -0.202288 | 0.109903 | 0.042075 | -0.221141 | -0.069408 | -0.205654 | -0.496180 | 0.205633 | 0.093595 | 1.000000 | 0.476166 |
| quality | 0.124052 | -0.390558 | 0.226373 | 0.013732 | -0.128907 | -0.050656 | -0.185100 | -0.174919 | -0.057731 | 0.251397 | 0.476166 | 1.000000 |
plt.figure(figsize=(15,10))
sns.heatmap(red.corr(), annot=True, cmap='coolwarm')
plt.show()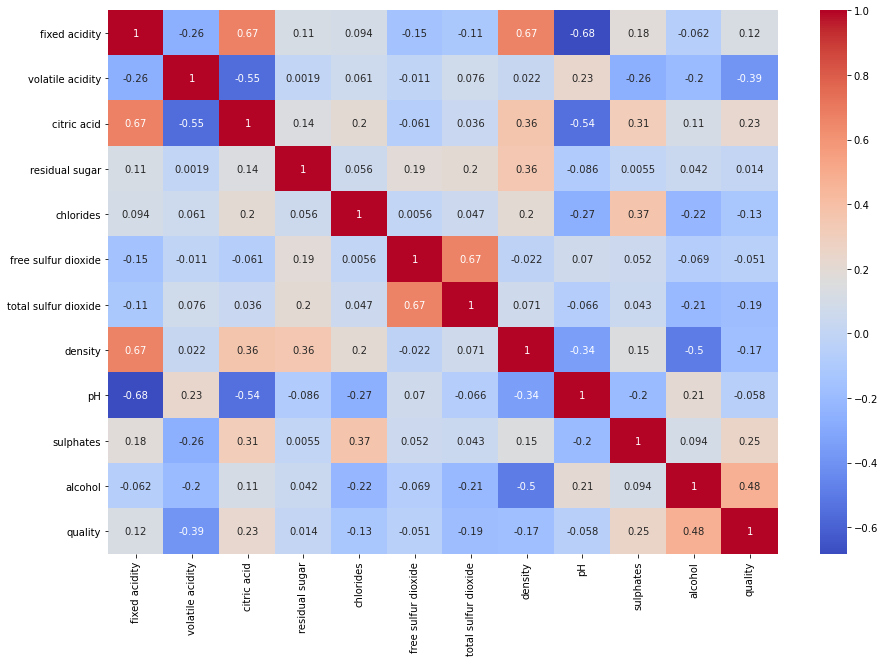
Existen correlaciones positivas entre las características:
fixed acidityconcitric acidydensidad,free sulfur dioxidecontotal sulfur dioxide,alcoholconquality
y correlaciones negativas entre las caracteríticas:
fixed acidityconpH,volatile acidityconcitric acid,citric acidconpH,densityconalcohol
Mostremos sólo las correlaciones de la variable objetivo con el resto de características:
corr_matrix["quality"].drop("quality").sort_values(ascending=False)alcohol 0.476166
sulphates 0.251397
citric acid 0.226373
fixed acidity 0.124052
residual sugar 0.013732
free sulfur dioxide -0.050656
pH -0.057731
chlorides -0.128907
density -0.174919
total sulfur dioxide -0.185100
volatile acidity -0.390558
Name: quality, dtype: float64
plt.figure(figsize=(8,5))
corr_matrix["quality"].drop("quality").sort_values(ascending=False).plot(kind='bar')
plt.title("Attribute correlations with quality")
plt.show()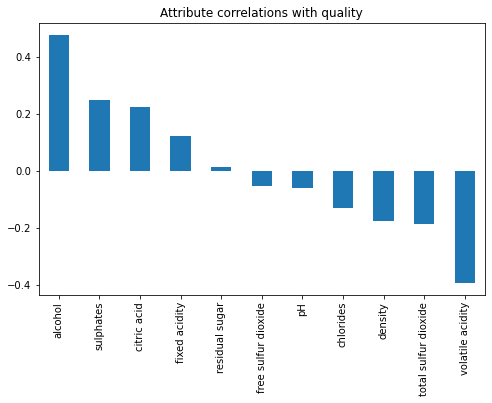
Podemos observar una correlación positiva con el atributo alcohol y negativa con volatile acidity.
Preparación de los datos
En primer lugar vamos a crear el conjunto de predictores y el conjunto con la variable objetivo:
predict_columns = red.columns[:-1]
predict_columnsIndex(['fixed acidity', 'volatile acidity', 'citric acid', 'residual sugar',
'chlorides', 'free sulfur dioxide', 'total sulfur dioxide', 'density',
'pH', 'sulphates', 'alcohol'],
dtype='object')
X = red[predict_columns]
y = red["quality"]Posteriormente, creamos los conjuntos de entrenamiento y prueba, siendo el conjunto de entrenamiento un 80% del dataset completo y el 20% restante el conjunto de prueba:
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42, test_size=0.2)X_train.shape, y_train.shape((1279, 11), (1279,))
X_test.shape, y_test.shape((320, 11), (320,))
Línea base
Para determinar adecuadamente si nuestro modelo es mejor o peor, primero tenemos que definir una línea base con la que poder comparar. Para ello vamos a entrenar algunos regresores dummy cuyos resultados usaremos como línea base de comparación.
Este regresor dummy predice de manera constante la puntuación 5, la más frecuente:
rg_dummy = DummyRegressor(strategy="constant", constant=5)
rg_dummy.fit(X_train, y_train)DummyRegressor(constant=array(5), strategy='constant')
Nos creamos una función que nos permitirá evaluar nuestro modelo a lo largo de este análisis:
def evaluate_model(estimator, X_train, y_train, cv=10, verbose=True):
"""Print and return cross validation of model
"""
scoring = ["neg_mean_absolute_error", "neg_mean_squared_error", "r2"]
scores = cross_validate(estimator, X_train, y_train, return_train_score=True, cv=cv, scoring=scoring)
val_mae_mean, val_mae_std = -scores['test_neg_mean_absolute_error'].mean(), \
-scores['test_neg_mean_absolute_error'].std()
train_mae_mean, train_mae_std = -scores['train_neg_mean_absolute_error'].mean(), \
-scores['train_neg_mean_absolute_error'].std()
val_mse_mean, val_mse_std = -scores['test_neg_mean_squared_error'].mean(), \
-scores['test_neg_mean_squared_error'].std()
train_mse_mean, train_mse_std = -scores['train_neg_mean_squared_error'].mean(), \
-scores['train_neg_mean_squared_error'].std()
val_rmse_mean, val_rmse_std = np.sqrt(-scores['test_neg_mean_squared_error']).mean(), \
np.sqrt(-scores['test_neg_mean_squared_error']).std()
train_rmse_mean, train_rmse_std = np.sqrt(-scores['train_neg_mean_squared_error']).mean(), \
np.sqrt(-scores['train_neg_mean_squared_error']).std()
val_r2_mean, val_r2_std = scores['test_r2'].mean(), scores['test_r2'].std()
train_r2_mean, train_r2_std = scores['train_r2'].mean(), scores['train_r2'].std()
result = {
"Val MAE": val_mae_mean,
"Val MAE std": val_mae_std,
"Train MAE": train_mae_mean,
"Train MAE std": train_mae_std,
"Val MSE": val_mse_mean,
"Val MSE std": val_mse_std,
"Train MSE": train_mse_mean,
"Train MSE std": train_mse_std,
"Val RMSE": val_rmse_mean,
"Val RMSE std": val_rmse_std,
"Train RMSE": train_rmse_mean,
"Train RMSE std": train_rmse_std,
"Val R2": val_r2_mean,
"Val R2 std": val_r2_std,
"Train R2": train_rmse_mean,
"Train R2 std": train_r2_std,
}
if verbose:
print(f"val_MAE_mean: {val_mae_mean} - (std: {val_mae_std})")
print(f"train_MAE_mean: {train_mae_mean} - (std: {train_mae_std})")
print(f"val_MSE_mean: {val_mse_mean} - (std: {val_mse_std})")
print(f"train_MSE_mean: {train_mse_mean} - (std: {train_mse_std})")
print(f"val_RMSE_mean: {val_rmse_mean} - (std: {val_rmse_std})")
print(f"train_RMSE_mean: {train_rmse_mean} - (std: {train_rmse_std})")
print(f"val_R2_mean: {val_r2_mean} - (std: {val_r2_std})")
print(f"train_R2_mean: {train_r2_mean} - (std: {train_r2_std})")
return resultrg_scores = evaluate_model(rg_dummy, X_train, y_train)val_MAE_mean: 0.719365157480315 - (std: -0.06352462970037416)
train_MAE_mean: 0.7193126146346173 - (std: -0.007057414168822716)
val_MSE_mean: 1.0398868110236221 - (std: -0.12176257291946108)
train_MSE_mean: 1.0398750482672072 - (std: -0.01354074583910719)
val_RMSE_mean: 1.0180017820772593 - (std: 0.05965888627141756)
train_RMSE_mean: 1.0197209977802941 - (std: 0.006643414270421584)
val_R2_mean: -0.6192850555554466 - (std: 0.14799333040101653)
train_R2_mean: -0.5986022943608599 - (std: 0.01598456942915052)
Un regresor que siempre predice la puntuación de calidad más frecuente (en nuestro caso, la puntuación 5) obtiene un RMSE = 1.01.
Probemos ahora con un regresor dummy que predice la media de las puntuaciones de calidad:
rg_dummy = DummyRegressor(strategy="mean") # Mean prediction
rg_dummy.fit(X_train, y_train)DummyRegressor()
rg_scores = evaluate_model(rg_dummy, X_train, y_train)val_MAE_mean: 0.6842639509806605 - (std: -0.039939453843720794)
train_MAE_mean: 0.6836374055181736 - (std: -0.004461928774514038)
val_MSE_mean: 0.6515564887161005 - (std: -0.08938937463665708)
train_MSE_mean: 0.6505431870574859 - (std: -0.009928873673332832)
val_RMSE_mean: 0.8052590895459458 - (std: 0.05580580095057208)
train_RMSE_mean: 0.8065390950374436 - (std: 0.006154285796714715)
val_R2_mean: -0.007632943779434287 - (std: 0.010684535533448955)
train_R2_mean: 0.0 - (std: 0.0)
Un regresor que predice siempre la puntuación media de calidad obtiene un RMSE = 0.80. Vamos a tomar la predicción de este regresor dummy como nuestra línea base.
Entrenamiento de diversos modelos
OK, ya estamos listos para entrenar varios modelos de forma rápida de diferente tipología y usando los parámetros estándar. Seleccionamos algunos modelos de regresión: Linear Regression, Lasso, ElasticNet, Ridge, Extre Trees, y RandomForest.
models = [LinearRegression(), Lasso(alpha=0.1), ElasticNet(),
Ridge(), ExtraTreesRegressor(), RandomForestRegressor()]
model_names = ["Lineal Regression", "Lasso", "ElasticNet",
"Ridge", "Extra Tree", "Random Forest"]mae = []
mse = []
rmse = []
r2 = []
for model in range(len(models)):
print(f"Paso {model+1} de {len(models)}")
print(f"...running {model_names[model]}")
rg_scores = evaluate_model(models[model], X_train, y_train)
mae.append(rg_scores["Val MAE"])
mse.append(rg_scores["Val MSE"])
rmse.append(rg_scores["Val RMSE"])
r2.append(rg_scores["Val R2"])Paso 1 de 6
...running Lineal Regression
val_MAE_mean: 0.5054157041773433 - (std: -0.046264972549372924)
train_MAE_mean: 0.49951141240221786 - (std: -0.005396834677886112)
val_MSE_mean: 0.4363366846653876 - (std: -0.0713599197838867)
train_MSE_mean: 0.423559916011364 - (std: -0.007783364048942027)
val_RMSE_mean: 0.6578988186927084 - (std: 0.059210041615646476)
train_RMSE_mean: 0.6507877560250832 - (std: 0.005934022177307515)
val_R2_mean: 0.32302131635332426 - (std: 0.0972958323285871)
train_R2_mean: 0.34888336017832816 - (std: 0.008988207786517072)
Paso 2 de 6
...running Lasso
val_MAE_mean: 0.5542159398138832 - (std: -0.044044881537899525)
train_MAE_mean: 0.551926769360105 - (std: -0.005222359881914205)
val_MSE_mean: 0.5011613158962728 - (std: -0.07980261731926688)
train_MSE_mean: 0.49648903729654775 - (std: -0.00886434349442919)
val_RMSE_mean: 0.7054560563903938 - (std: 0.05910218607112876)
train_RMSE_mean: 0.7045920060170998 - (std: 0.006256385006291075)
val_R2_mean: 0.22550457016915199 - (std: 0.06858817248045986)
train_R2_mean: 0.23679715721911138 - (std: 0.008061051196907644)
Paso 3 de 6
...running ElasticNet
val_MAE_mean: 0.6484828644185054 - (std: -0.03858618665902155)
train_MAE_mean: 0.6472074434172257 - (std: -0.004861676284701619)
val_MSE_mean: 0.6260699925252777 - (std: -0.08837053843631361)
train_MSE_mean: 0.6236958050351286 - (std: -0.009753039023728842)
val_RMSE_mean: 0.7891968495348196 - (std: 0.056906284447264595)
train_RMSE_mean: 0.7897200517246066 - (std: 0.0061680579774354895)
val_R2_mean: 0.032300440343033296 - (std: 0.027013749786509673)
train_R2_mean: 0.041268269123349036 - (std: 0.0034334107542665303)
Paso 4 de 6
...running Ridge
val_MAE_mean: 0.5052017417711606 - (std: -0.04639189777979148)
train_MAE_mean: 0.5000120146851917 - (std: -0.00538293390792397)
val_MSE_mean: 0.4353611411950837 - (std: -0.07150445371257734)
train_MSE_mean: 0.4243933932521361 - (std: -0.007774091981744382)
val_RMSE_mean: 0.6571341500690723 - (std: 0.05946301378236467)
train_RMSE_mean: 0.6514279204128516 - (std: 0.0059209592739344254)
val_R2_mean: 0.32476443307512515 - (std: 0.09605257129964452)
train_R2_mean: 0.3476024511130947 - (std: 0.0089301257345918)
Paso 5 de 6
...running Extra Tree
val_MAE_mean: 0.3767233021653543 - (std: -0.048411131876621855)
train_MAE_mean: -0.0 - (std: -0.0)
val_MSE_mean: 0.33849758981299216 - (std: -0.07037684927470149)
train_MSE_mean: -0.0 - (std: -0.0)
val_RMSE_mean: 0.5784725891678845 - (std: 0.062185636560190514)
train_RMSE_mean: 0.0 - (std: 0.0)
val_R2_mean: 0.4753582472917177 - (std: 0.09435328966382882)
train_R2_mean: 1.0 - (std: 0.0)
Paso 6 de 6
...running Random Forest
val_MAE_mean: 0.421939406988189 - (std: -0.03848180259232641)
train_MAE_mean: 0.15720688154624 - (std: -0.0024955091475250693)
val_MSE_mean: 0.3536394728100393 - (std: -0.06315688035394738)
train_MSE_mean: 0.049982221460505356 - (std: -0.0012897300801719821)
val_RMSE_mean: 0.5921558699969636 - (std: 0.05468910712544724)
train_RMSE_mean: 0.22354850027354933 - (std: 0.0028791467403151403)
val_R2_mean: 0.450229047801475 - (std: 0.08970981370698214)
train_R2_mean: 0.9231573754360927 - (std: 0.0020715859571618753)
Veamos cuál es el rendimiento de cada uno de ellos:
df_result = pd.DataFrame({"Model": model_names,
"MAE": mae,
"MSE": mse,
"RMSE": rmse,
"R2": r2})
df_result| Model | MAE | MSE | RMSE | R2 | |
|---|---|---|---|---|---|
| 0 | Lineal Regression | 0.505416 | 0.436337 | 0.657899 | 0.323021 |
| 1 | Lasso | 0.554216 | 0.501161 | 0.705456 | 0.225505 |
| 2 | ElasticNet | 0.648483 | 0.626070 | 0.789197 | 0.032300 |
| 3 | Ridge | 0.505202 | 0.435361 | 0.657134 | 0.324764 |
| 4 | Extra Tree | 0.375012 | 0.335985 | 0.576599 | 0.479648 |
| 5 | Random Forest | 0.422140 | 0.356897 | 0.594764 | 0.445618 |
df_result.sort_values(by="RMSE", ascending=False).plot.barh("Model", "RMSE");
df_result.sort_values(by="R2").plot.barh("Model", "R2");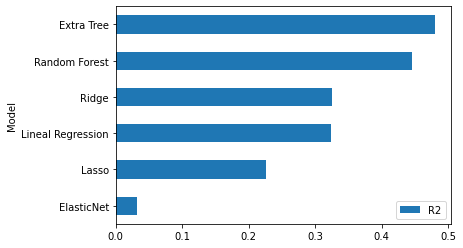
Analizando los resultados vemos que extra trees es el modelo que mejores resultados obtiene. RMSE = 0.576599 and R2 = 0.479648. OK, este será nuestro modelo candidato. Vamos a realizar el ajuste fino.
Fine-Tune
param_grid = [
{'n_estimators': range(10, 300, 10), 'max_features': [2, 3, 4, 5, 8, "auto"], 'bootstrap': [True, False]}
]
xtree_reg = ExtraTreesRegressor(random_state=42, n_jobs=-1)
grid_search = GridSearchCV(xtree_reg, param_grid, cv=5,
scoring='neg_mean_squared_error',
return_train_score=True)
grid_search.fit(X_train, y_train)GridSearchCV(cv=5, estimator=ExtraTreesRegressor(n_jobs=-1, random_state=42),
param_grid=[{'bootstrap': [True, False],
'max_features': [2, 3, 4, 5, 8, 'auto'],
'n_estimators': range(10, 300, 10)}],
return_train_score=True, scoring='neg_mean_squared_error')
grid_search.best_params_{'bootstrap': False, 'max_features': 5, 'n_estimators': 160}
¡Es el momento de la verdad! Veamos su rendimiento en el conjunto de prueba:
final_model = grid_search.best_estimator_
y_pred = final_model.predict(X_test)print(f"MAE: {metrics.mean_absolute_error(y_test, y_pred)}")
print(f"MSE: {metrics.mean_squared_error(y_test, y_pred)}")
print(f"RMSE: {np.sqrt(metrics.mean_squared_error(y_test, y_pred))}")
print(f"R2: {final_model.score(X_test, y_test)}")MAE: 0.38298828124999995
MSE: 0.28038391113281247
RMSE: 0.5295128998738486
R2: 0.5709542506612473
Bueno, ¡un poco mejor! Obtenemos un error de +/- 0.5295.
Podemos visualizar cómo han sido sus predicciones:
plt.figure(figsize=(10,8))
plt.scatter(y_test, y_pred, alpha=0.1)
plt.xlabel("Real")
plt.ylabel("Predicted")
plt.show()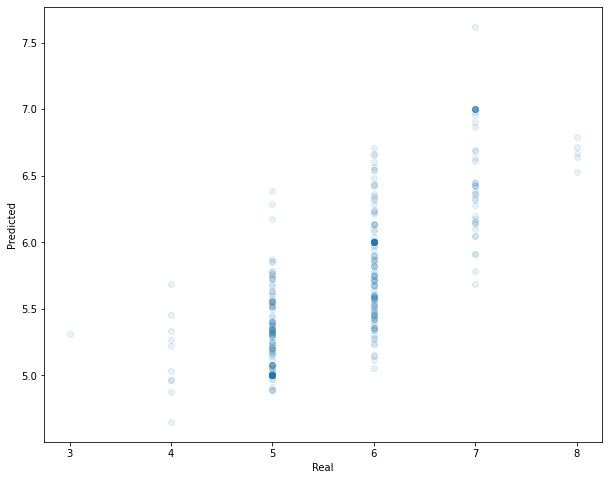
Se observa una mayor concentración de predicciones en las puntuaciones centrales (5 y 6), debido a un mayor número de instancias en el dataset respecto a las demás. También podemos comprobar que las predicciones sobre las puntuaciones extremas son pésimas. Las puntuaciones 5 y 6 son las que mejores resultados ofrecen.
¿Cuáles son las características más relevantes?:
feature_importances = final_model.feature_importances_
feature_importancesarray([0.06242878, 0.12054219, 0.07478461, 0.06697772, 0.06670251,
0.05944129, 0.07925392, 0.07148382, 0.06178626, 0.12593217,
0.21066673])
sorted(zip(feature_importances, X_test.columns), reverse=True)[(0.2106667292131454, 'alcohol'),
(0.12593217102849735, 'sulphates'),
(0.1205421943281732, 'volatile acidity'),
(0.07925392046422035, 'total sulfur dioxide'),
(0.07478461494308856, 'citric acid'),
(0.07148382305429932, 'density'),
(0.06697771630809285, 'residual sugar'),
(0.06670250522733821, 'chlorides'),
(0.062428775664599805, 'fixed acidity'),
(0.061786258397281676, 'pH'),
(0.05944129137126337, 'free sulfur dioxide')]
feature_imp = pd.Series(feature_importances, index=X_train.columns).sort_values(ascending=False)
feature_imp.plot(kind='bar')
plt.title('Feature Importances')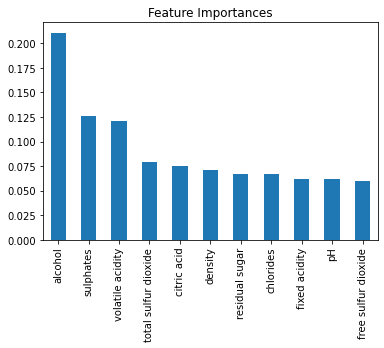
La gráfica nos muestra que las características más importantes son: alcohol, sulphates y volatile acidity, algo que también nos anticipaba el análisis de correlaciones que vimos anteriormente.
Veamos ahora cómo se distribuyen los errores:
df_resul = pd.DataFrame({"Pred": y_pred,
"Real": y_test,
"error": y_pred - y_test,
"error_abs": abs(y_pred - y_test)})df_resul["error"].plot.hist(bins=40, density=True)
plt.title("Error distribution")
plt.xlabel("Error");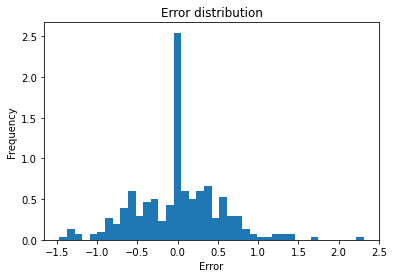
Parece que los errores siguen una distribución gaussiana.
¿Cuál es el MAE que se produce en la puntuación de calidad 6?
df_resul[df_resul["Real"].isin([6])]["error"].abs().mean()0.3437013037105609
Más en general ¿Cuál es el MAE que se produce en cada puntuación de calidad?
df_resul.groupby("Real")["error_abs"].mean()Real
3 2.268966
4 1.286657
5 0.462774
6 0.343701
7 0.617315
8 1.597190
9 3.434483
Name: error_abs, dtype: float64
df_resul.groupby("Real")["error_abs"].mean().plot.bar()
plt.title("MAE distribution")
plt.ylabel("MAE")
plt.xlabel("Quality");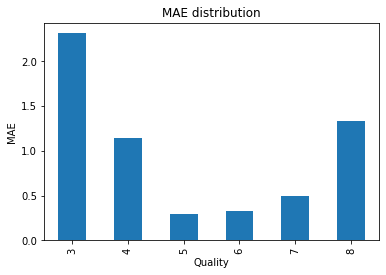
Se comprueba que en las puntuaciones de calidad extremas el error es elevado, sobre todo en la puntuación 8 y 3. Las puntuaciones 5 y 6 es donde menos error se produce.
Guardado del modelo
Como paso final, guardamos nuestro modelo entrenado para futuras predicciones.
import joblib
joblib.dump(final_model, "final_model.joblib", compress=True)['final_model.joblib']
Conclusiones
Después de probar diversos modelos, el que mejores resultados arroja es ExtraTrees. Tras un ajuste fino del mismo conseguimos una importante mejora:
- Nuestra línea base teníamos un MAE: 0.684263 y RMSE: 0.805259.
- El modelo de Extra Tree obtiene un MAE: 0.382988, RMSE: 0.529512 y R2:0.570954.
Sin embargo, la puntuación de R2 sigue siendo muy baja. Según dicho valor, nuestro modelo apenas puede explicar un 57% de la varianza. Es decir, el porcentaje de relación entre las variables que puede explicarse mediante nuestro modelo lineal es del 57,09%.
Como sabemos, R2 varía entre 0 y 1. Es la proporción de la varianza en la variable dependiente (nuestra variable objetivo) que es predecible a partir de las variables independientes (nuestros predictores). Si la predicción fuera exactamente igual a lo real, R2 = 1 (es decir, 100%).
El RMSE = 0,529. Es decir, tenemos un error típico de predicción de 0,529.
Según la gráfica de distribución de MAE podemos observar que nuestro modelo no es nada bueno para valores extremos de puntuación. De hecho no es capaz de predecir ninguna puntuación de 3 y apenas alguna de 8. Según vimos en la distribución de la variable objetivo, ésta se encuentra muy desbalanceada, apenas existen observaciones para los valores extremos, por lo que el modelo no tiene suficientes datos de entrenamiento para todas las puntuaciones de calidad.
Como ejercicio final, podríamos probar a enfocar el modelado como un problema de clasificación, para evaluar si ofrece mejores resultados que un problema de regresión. Lo veremos en futuros posts.